誰でもできるRNA-seq解析シリーズ!
今回はHISAT2によるマッピング結果をアセンブルし、発現差を比較するようにします。
StringTieというソフトを使用します。
↓これまでの関連記事一覧↓
lifesciencehack-ai.hatenablog.com
sam→bamへの変換
まず、アノテーションを実施する前に、
HISAT2でマッピングしたファイルをソートしてバイナリファイルのbam形式に変換します。
こうすることによってコンピュータが認識しやすくなり、
高速に処理することができるそうです。
ソート&bam変換は「samtools」を使います。
samtoolsのインストール
まずは、samtoolsをインストールします。
インストールはbrew installで簡単にできます。
brew install samtools
samファイルをソートしてbamに変換
samtools sort -@ 4 -O bam -o SRR1571967.sort.bam SRR1571967.sam
samtoolsのコマンド説明
samtools samtoolコマンド実行を指示
sort samtoolでソートする
-@ 4 CPUの使用するスレッド数を指定
-O bam outputのファイル形式をbamに指定
-o SRR1571967.sort.bam outputのファイル名を指定
SRR1571967.sam hisat2で出力したマッピングファイルを指定
すべてのファイルに対して同様に実施しましょう。
samtools sort -@ 4 -O bam -o SRR1571967.sort.bam SRR1571967.sam samtools sort -@ 4 -O bam -o SRR1571968.sort.bam SRR1571968.sam samtools sort -@ 4 -O bam -o SRR1571969.sort.bam SRR1571969.sam samtools sort -@ 4 -O bam -o SRR1571970.sort.bam SRR1571970.sam samtools sort -@ 4 -O bam -o SRR1571971.sort.bam SRR1571971.sam samtools sort -@ 4 -O bam -o SRR1571972.sort.bam SRR1571972.sam
これで全てのマッピングファイルをソートしbamに変換できました。
StringTieでassembleする
マッピング→ソート→bam変換が完了しました。
今あるbamデータはRNA-seqの何千万とあるリードが、
ゲノムのどこに相当するものかという情報だけです。
これからこのデータをもとに遺伝子発現量を比較するわけですが、
そのためにはゲノムのどこに何の遺伝子があるのかを知り、
各リードは何の遺伝子の一部であったのか情報を付与する必要があります。
このためのプログラムが「StringTie」になります。
StringTieのインストール
以下のコマンドを実行
brew install stringtie
アノテーションファイルの取得
次にアノテーションファイルを取得いたします。
リファレンスゲノムと同じデータベースからアノテーションを取ります。
今回はUCSCのmm10をリファレンスゲノムとしましたので、
UCSCからアノテーションファイルを取得します。
UCSCのTable Browserに行き、対応するアノテーションファイルを取得します。
Table Browser
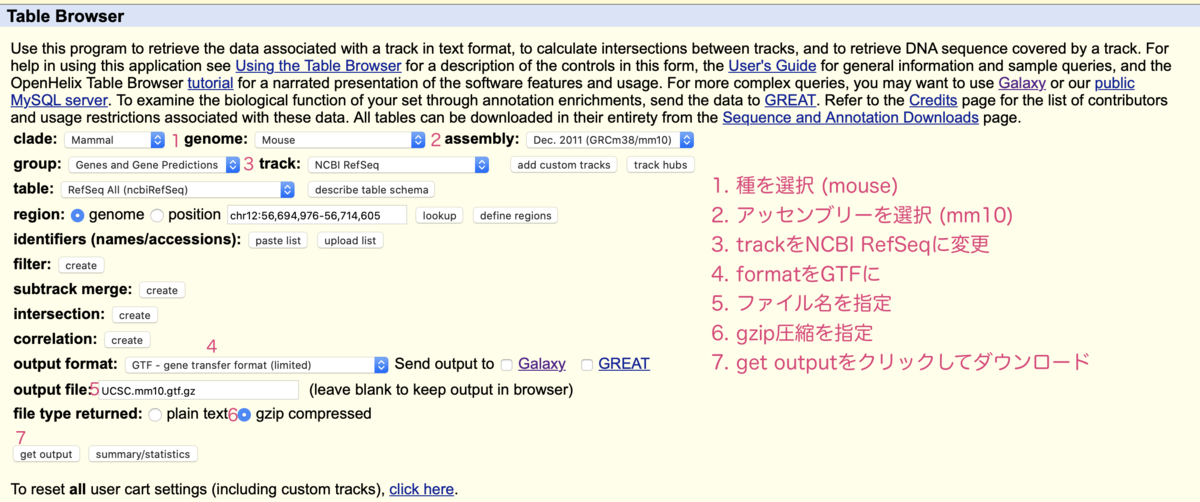 画像のように、
画像のように、
●種
●リファレンスゲノムに対応するアッセンブリー
●TrackをNCBI RefSeq
●gtfファイル形式を選択
●ファイル名指定
●gzip圧縮
を設定しget outputをクリックしてダウンロードします。
macOSですと、勝手にファイルが解凍されると思います。
もし解凍されていなかったら、以下のコマンドで解凍しましょう。
#gzipファイルの解凍 gzip -d UCSC.mm10.gtf.gz
StringTieによるアノテーション情報付与
stringtie SRR1571967.sort.bam -o SRR1571967.gtf -p 4 -G UCSC.mm10.gtf -l SRR1571967 -A SRR1571967.gene.abundance.tab
StringTieコマンド説明
stringtie stringtieコマンド実行
SRR1571967.sort.bam samtoolsでsortしたbamファイルを指定
-o SRR1571967.gtf出力ファイル名を指定
-p 4CPUの使用するスレッド数を指定
-G UCSC.mm10.gtf先程UCSCからダウンロードしたアノテーションファイルを指定
-l SRR1571967ラベル名を指定
-A SRR1571967.gene.abundance.tab タブ区切り形式ファイルも出力させ、そのファイル名を指定
StringTieの詳しい説明につきましては、
https://ccb.jhu.edu/software/stringtie/index.shtml?t=manual#output
に詳しい説明があります。
これを全てのsort.bamファイルに対して実行します。
stringtie SRR1571967.sort.bam -o SRR1571967.gtf -p 4 -G UCSC.mm10.gtf -l SRR1571967 -A SRR1571967.gene.abundance.tab stringtie SRR1571968.sort.bam -o SRR1571968.gtf -p 4 -G UCSC.mm10.gtf -l SRR1571968 -A SRR1571968.gene.abundance.tab stringtie SRR1571969.sort.bam -o SRR1571969.gtf -p 4 -G UCSC.mm10.gtf -l SRR1571969 -A SRR1571969.gene.abundance.tab stringtie SRR1571970.sort.bam -o SRR1571970.gtf -p 4 -G UCSC.mm10.gtf -l SRR1571970 -A SRR1571970.gene.abundance.tab stringtie SRR1571971.sort.bam -o SRR1571971.gtf -p 4 -G UCSC.mm10.gtf -l SRR1571971 -A SRR1571971.gene.abundance.tab stringtie SRR1571972.sort.bam -o SRR1571972.gtf -p 4 -G UCSC.mm10.gtf -l SRR1571972 -A SRR1571972.gene.abundance.tab
実行すると作業ディレクトリに下画像のようにファイルが作成されたらOKです。
gtfファイルをmergeする
次にStringTieで作成された.gtfファイルを一つにまとめ、比較できるようにします。
各サンプルで同定された転写産物をまとめたgtfファイルが作成されます。
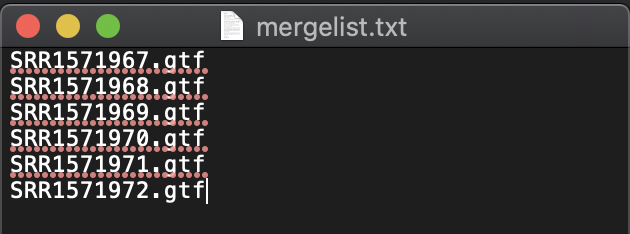
mergelist.txtファイルの作成
mergeさせるgtfファイル(stringtieでアノテーションをつけた)のpathを全て指定するtxtファイルを作成します。
一番ラクなのは全てのファイルを作業ディレクトリに入れておけば、
画像のようにファイル名一覧を作成するだけで良いです。
macであればテキストエディットで作成し、ファイル名をmergelist.txtにして保存しましょう。
作業ディレクトリにファイルがない場合は相対パスを一つづつ書けばOKです。
mergeの実行
stringtie --merge -G UCSC.mm10.gtf -o stringtie_merged.gtf mergelist.txt
コマンドの説明
stringtie --mergestringtieでmergeを実行することを指定
G UCSC.mm10.gtf リファレンスゲノムを指定
-o stringtie_merged.gtf mergelist.txt 出力されるファイル名を指定、mergeするファイルを指定したtxtファイル名を指定
mergeを実行し、stringtie_merged.gtfファイルが作成されればOKです。
Ballgown用ファイルの作成
作業ディレクトリにballgownというフォルダを作ります。
その後、
stringtie SRR1571967.sort.bam -e -B -p 4 -G stringtie_merged.gtf -o ballgown/SRR1571967/SRR1571967.gtf
コマンド説明
stringtiestringtieを実行
SRR1571967.sort.bam sort.bamファイルを指定
-e -Gで指定されたリファレンス転写物にマッチした転写物のみを使用するオプション
-B Ballgown用ファイルを作成
-p 4 使用するCPUのスレッド数を指定
-G stringtie_merged.gtf 先ほどStringtieでmergeしたファイルを指定
-o ballgown/SRR1571967/SRR1571967.gtf 出力先とファイル名を指定
全てのファイルに対して実行
stringtie SRR1571967.sort.bam -e -B -p 4 -G stringtie_merged.gtf -o ballgown/SRR1571967/SRR1571967.gtf stringtie SRR1571968.sort.bam -e -B -p 4 -G stringtie_merged.gtf -o ballgown/SRR1571968/SRR1571968.gtf stringtie SRR1571969.sort.bam -e -B -p 4 -G stringtie_merged.gtf -o ballgown/SRR1571969/SRR1571969.gtf stringtie SRR1571970.sort.bam -e -B -p 4 -G stringtie_merged.gtf -o ballgown/SRR1571970/SRR1571970.gtf stringtie SRR1571971.sort.bam -e -B -p 4 -G stringtie_merged.gtf -o ballgown/SRR1571971/SRR1571971.gtf stringtie SRR1571972.sort.bam -e -B -p 4 -G stringtie_merged.gtf -o ballgown/SRR1571972/SRR1571972.gtf
次の記事ではballgownを用いて発現差解析を行っていきたいと思います。